![]()
O estudo, ontem publicado na revista científica Nature Communications, foi desenvolvido a partir de técnicas computacionais, com as quais a equipa do MIT comparou o genoma do novo coronavírus, que causa a Covid-19, com o genoma de outros coronavírus similares, como o SARS-CoV (que causa a Síndrome Respiratória Aguda Grave).
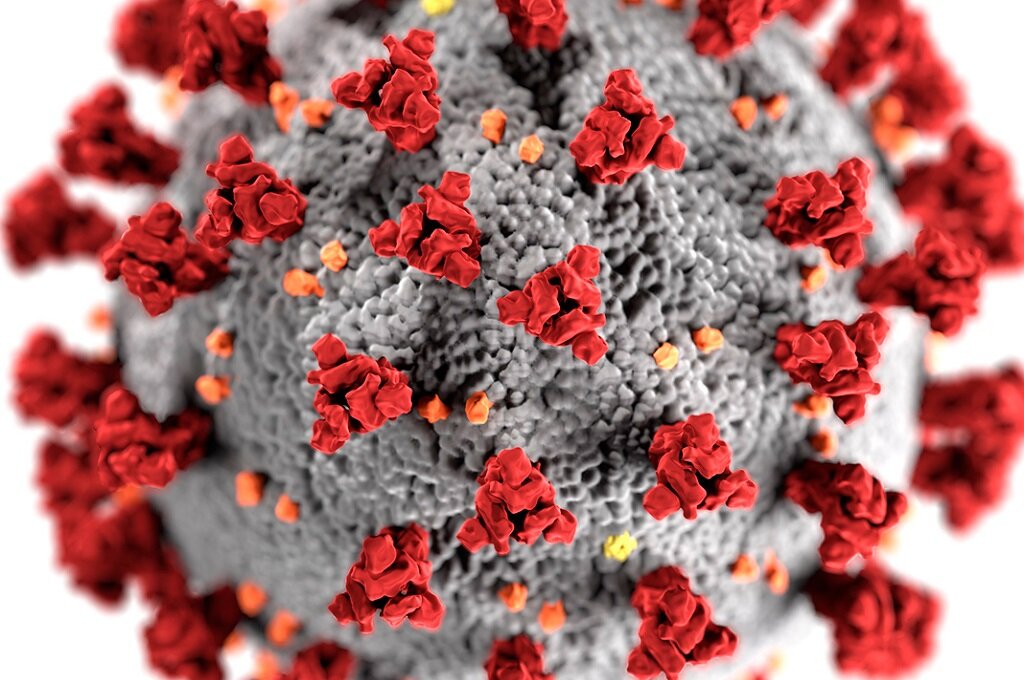
O genoma define-se, em termos latos, por toda a informação genética dada por um conjunto de genes formados por sequências de moléculas de ADN ou ARN e que codificam determinadas proteínas.
Para este trabalho, os investigadores do MIT determinaram o conjunto de genes codificadores de proteínas do SARS-CoV-2 e analisaram a probabilidade de novas mutações (alterações no material genético) ajudarem o novo coronavírus a adaptar-se aos humanos e a tornar-se mais contagioso ou escapar ao sistema imunológico.
A equipa confirmou seis genes codificadores de proteínas no genoma do SARS-CoV-2, além dos cinco comuns para os coronavírus, mas descobriu que outros supostos genes, afinal, não codificam nenhuma proteína.
O estudo analisou também mais de 1.800 mutações genéticas que surgiram no SARS-CoV-2 desde que foi identificado pela primeira vez.
Para cada gene, os cientistas compararam a rapidez com que evoluiu no passado e desde o início da pandemia da Covid-19.
Descobriram, por exemplo, que uma região da proteína do nucleocapsídeo, que envolve e protege o material genético viral, no caso moléculas de ARN, tinha muito mais mutações do que era esperado para os seus padrões de evolução histórica.
Esta região é considerada um alvo para as células B humanas, células do sistema imunitário que produzem anticorpos (glicoproteínas que se ligam e neutralizam um antigénio como um vírus).
De acordo com os autores do estudo, mutações nesta região podem ajudar o SARS-CoV-2 a escapar ao sistema imunitário humano (e consequentemente à eficácia de vacinas, que induzem a produção de anticorpos no organismo).
“A região mais acelerada em todo o genoma do SARS-CoV-2 fica bem no meio da proteína do nucleocapsídeo. Supomos que as variantes que não sofrem mutações naquela região são reconhecidas pelo sistema imunitário humano e eliminadas, enquanto as variantes que acumulam mutações aleatoriamente naquela região são, na verdade, mais capazes de escapar ao sistema imunitário humano e permanecer em circulação”, afirmou, citado em comunicado do MIT, um dos autores da investigação, Manolis Kellis, que trabalha no Laboratório de Ciência Computacional e Inteligência Artificial do instituto.
Segundo a equipa do MIT que conduziu o estudo, os dados recolhidos podem ajudar outros cientistas a centrarem a sua atenção nas mutações genéticas do novo coronavírus que parecem ter efeitos significativos na sua capacidade infecciosa.
O SARS-CoV-2, que causa a doença respiratória Covid-19, foi detetado no final de 2019, em Wuhan, uma cidade do centro da China, e disseminou-se rapidamente pelo mundo.
LUSA/HN



0 Comments