No dia em que se assinala o Dia das Doenças Raras, a P-BIO promove uma iniciativa dedicada a estas doenças, que conta com o depoimento de várias figuras reconhecidas pelo seu trabalho na área da saúde.


No dia em que se assinala o Dia das Doenças Raras, a P-BIO promove uma iniciativa dedicada a estas doenças, que conta com o depoimento de várias figuras reconhecidas pelo seu trabalho na área da saúde.
Os casos de celulite necrotizante reapareceram “com tanta força” em 2023 em São Tomé e Princípe, infetando 350 pessoas, que a enfermaria do hospital do arquipélago está lotada com pacientes, disseram à Lusa fontes hospitalares.
Omjjara é o primeiro e único medicamento na UE indicado especificamente para o tratamento da esplenomegalia ou sintomas relacionados com a doença nesta população de doentes

a União das Associações das Doenças Raras (RD-Portugal) acaba de lançar uma campanha nas redes sociais, a decorrer ao longo do mês de dezembro, que tem como propósito sensibilizar para o facto de muitas doenças raras causarem deficiência
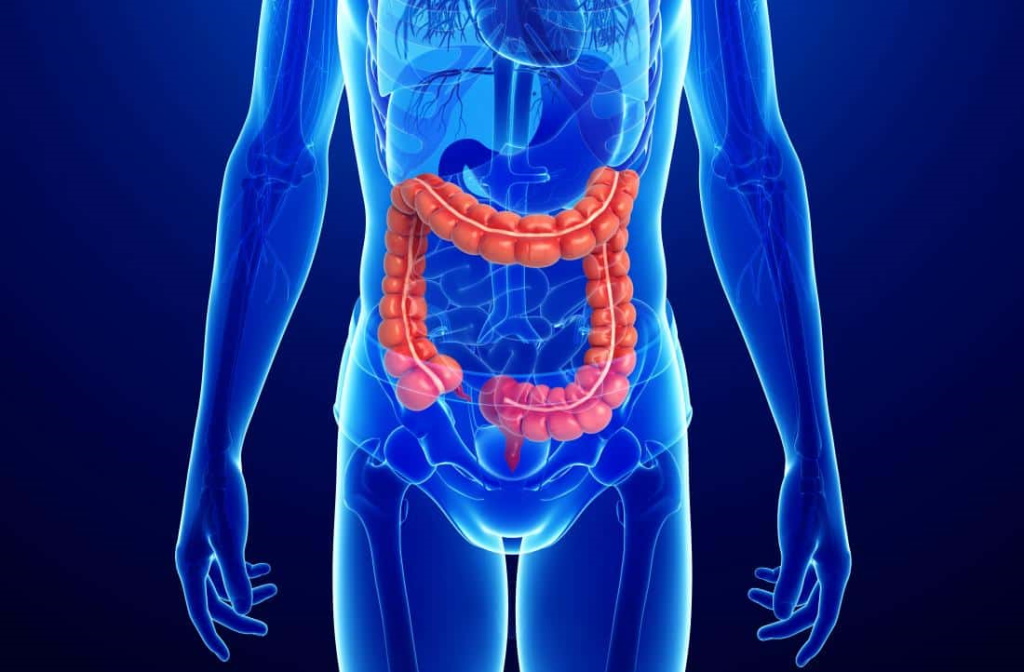
Um em cada quatro doentes de crohn inquiridos num estudo da Associação Portuguesa da Doença Inflamatória do Intestino sentiram grande impacto da pandemia na gestão da doença, que, nalguns casos, implica faltar, em média, quatro dias/mês ao trabalho

Susana Castro Marques
Diretora Médica da Pfizer Portugal

A Miocardiopatia amiloide associada a transtirretina (ATTR-CM) é considerada uma doença rara que afeta vários órgãos, em especial o coração. De acordo com o cardiologista do Centro Hospitalar de Lisboa Ocidental/ Hospital Santa Cruz, Carlos Aguiar, esta patologia...
A acromegalia é uma doença rara que se caracteriza pela produção excessiva da “hormona de crescimento”. O Diretor do Serviço de Endocrinologia, Diabetes e Metabolismo do Centro Hospitalar Universitário de São João esclarece que o quadro clínico em crianças e adolescentes se manifesta por “gigantismo”, em que o doente apresenta uma estatura elevada, enquanto em adultos a doença provoca anomalias nos tecidos moles, verificando-se um crescimento do nariz, orelhas, lábios, língua, entre outros. O diagnóstico é o principal desafio, podendo associar-se a complicações como diabetes, hipertensão e insuficiência cardíaca.

Na forma de apresentação neurológica os doentes costumam ter uma mistura de dores, às quais damos o nome de “dores nevrálgicas”, que têm características próprias porque são provocadas pela lesão do nervo periférico – é uma espécie de choque elétrico ou picadela. A perda de sensibilidade é outra forma de manifestação e começa nos pés e vai subindo
Investigadores do Instituto Francis Crick (Reino Unido), do NCI (National Cancer Institute, EUA) e da Universidade de Aalborg (Dinamarca), descobriram que a vitamina D estimula o crescimento de um...
A organização Médicos Sem Fronteiras (MSF) ofereceu assistência psicológica às famílias das 98 pessoas que perderam a vida no naufrágio de 07 de abril em Nampula, norte de Moçambique, avançou aquela entidade em comunicado.
Mais de metade dos adolescentes experimentaram álcool e um em cada cinco fumou recentemente cigarros eletrónicos, alertou hoje o Escritório Regional Europeu da Organização Mundial de Saúde (OMS), num relatório sobre hábitos de saúde.
A base da ambulância de emergência médica da Maia do INEM foi assaltada hoje, tendo sido roubados um computador e peças de fardamento do instituto, disse à Lusa fonte o Sindicato dos Técnicos de Emergência Pré-Hospitalar (STEPH).
A farmacêutica AstraZeneca totalizou 2.180 milhões de dólares (cerca de 2.031 milhões de euros) de lucro no primeiro trimestre, mais 20% do que no mesmo período do ano passado.
A Câmara dos Representantes do Arizona aprovou hoje a abolição de uma lei de 1864 que proíbe a quase totalidade dos abortos, considerada aplicável há duas semanas pelo Supremo Tribunal norte-americano.

Investigadores do Instituto Francis Crick (Reino Unido), do NCI (National Cancer Institute, EUA) e da Universidade de Aalborg (Dinamarca), descobriram que a vitamina D estimula o crescimento de um...

A organização Médicos Sem Fronteiras (MSF) ofereceu assistência psicológica às famílias das 98 pessoas que perderam a vida no naufrágio de 07 de abril em Nampula, norte de Moçambique, avançou aquela entidade em comunicado.

Mais de metade dos adolescentes experimentaram álcool e um em cada cinco fumou recentemente cigarros eletrónicos, alertou hoje o Escritório Regional Europeu da Organização Mundial de Saúde (OMS), num relatório sobre hábitos de saúde.

A base da ambulância de emergência médica da Maia do INEM foi assaltada hoje, tendo sido roubados um computador e peças de fardamento do instituto, disse à Lusa fonte o Sindicato dos Técnicos de Emergência Pré-Hospitalar (STEPH).

A farmacêutica AstraZeneca totalizou 2.180 milhões de dólares (cerca de 2.031 milhões de euros) de lucro no primeiro trimestre, mais 20% do que no mesmo período do ano passado.